
引子
被忽视的百亿损失
当一头猪因呼吸道疾病死亡时,背后可能是12种病毒+164种细菌的联合绞杀!2025年初,华中农业大学联合国家农业微生物实验室,在《mSystems》发布覆盖全国25省的研究,首次系统性地揭开猪呼吸道疾病的“病原江湖”,其中猪繁殖与呼吸综合征病毒(PRRSV)仍然是流行最广、丰度最高的病毒病原体。这项研究不仅填补了全国尺度上猪呼吸道病原体谱的空白,更为疫苗研发、疾病防控提供了关键数据支撑。
1、研究方法
全国采样与高通量测序
该研究在2022年10月至2023年10月期间,从全国25个省的猪场收集了111份因呼吸道疾病死亡的猪肺样本。样本根据猪年龄(以70天为界分为幼猪和老猪)和地理区域(东北、北方、东部、中部、南方、西南和西北中国)分组,最终形成23个测序池。研究采用宏转录组测序和宏基因组测序相结合的方法,对样本中的病毒、细菌和抗菌素耐药基因(ARGs)进行了全面鉴定。这种高通量测序方法能够在一次分析中捕获所有病原体信息,避免了传统检测方法的局限性。
2、核心结果
病猪肺部的“病原体全景图”
研究团队通过高通量测序与生物信息学分析,从病猪肺部鉴定出病毒、细菌及耐药基因(ARGs)三大类关键生物因子,其种类和分布特征颠覆了对猪呼吸道疾病病因的传统认知。
2.1、21种病毒现身,既有“老熟人”也有“新威胁”
研究共鉴定出21种病毒(分属12个科),包括7种RNA病毒和14种DNA病毒。其中,既有已知的“主流致病病毒”,也有此前被忽视的新兴病毒:
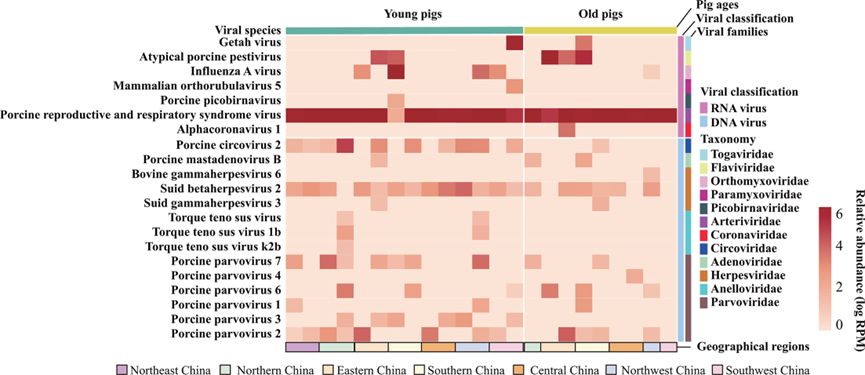
图1病毒组热图
通过复转录测序和宏基因组测序从中国患有呼吸系统疾病的猪肺组织中鉴定出的病毒种类。热图显示了从中国不同地区收集的猪肺部不同病毒物种的相对丰度,相对丰度值表示为每百万分对数(RPM)。
2.1.1、“头号元凶”PRRSV:分布最广、丰度最高
猪繁殖与呼吸综合征病毒(PRRSV)是本次研究中检出率最高的病毒——不仅在全国各地区病猪肺部均有发现,且在幼猪和成年猪中均保持高丰度。进一步分析显示,这些PRRSV均属于PRRSV2型(我国主流型别),主要分为4个谱系(L1、L3、L5、L8),其中L1谱系占绝对主导,代表菌株为近年来新兴的NADC-30和NADC-34。这一结果提示,当前我国猪呼吸道疾病的病毒感染中,PRRSV2型L1谱系是重点防控对象。
2.2.2、黑马现身:盖塔病毒(GETV)与新型猪呼吸道冠状病毒(PRCoV)
研究在华南地区病猪肺部首次检出盖塔病毒(GETV),且均属于Group III型。GETV是一种mosquito-borne人兽共患病病毒,此前已知会导致仔猪腹泻、母猪流产,本次发现其存在于呼吸道组织中,暗示它可能参与猪呼吸道疾病的发生,需警惕其跨物种传播风险。
此外,团队在华东地区成年病猪肺部发现一种α冠状病毒(HDD2株),与已知的猪呼吸道冠状病毒(PRCoV)高度同源。分子对接分析表明,该病毒刺突蛋白(Spike蛋白)能与猪呼吸道受体ACE2(ΔG=-24.7 kcal/mol)和肠道受体APN(ΔG=-40.4 kcal/mol)高效结合——这意味着该病毒不仅可能引发呼吸道感染,还可能具备肠道致病性,是一种“双靶点”潜在致病病毒。
2.2.3、潜伏势力:多型并存,感染复杂
猪圆环病毒(PCV)中,PCV2型是唯一在肺组织中检出的型别,但回顾性分析显示,PCV3型在2022年后流行率显著上升,PCV4型也已在我国猪场出现(需持续监测);流感病毒(IAV)以H1N1为主(2009年后成为主流),但H3N8、H1N5甚至H7N9等型别也有检出,提示流感病毒的遗传多样性;猪细小病毒(PPV)则检出6种型别(PPV1~PPV4、PPV6~PPV7),其中PPV1在近年样本中占比最高,此外,疱疹病毒、腺病毒也较为常见。
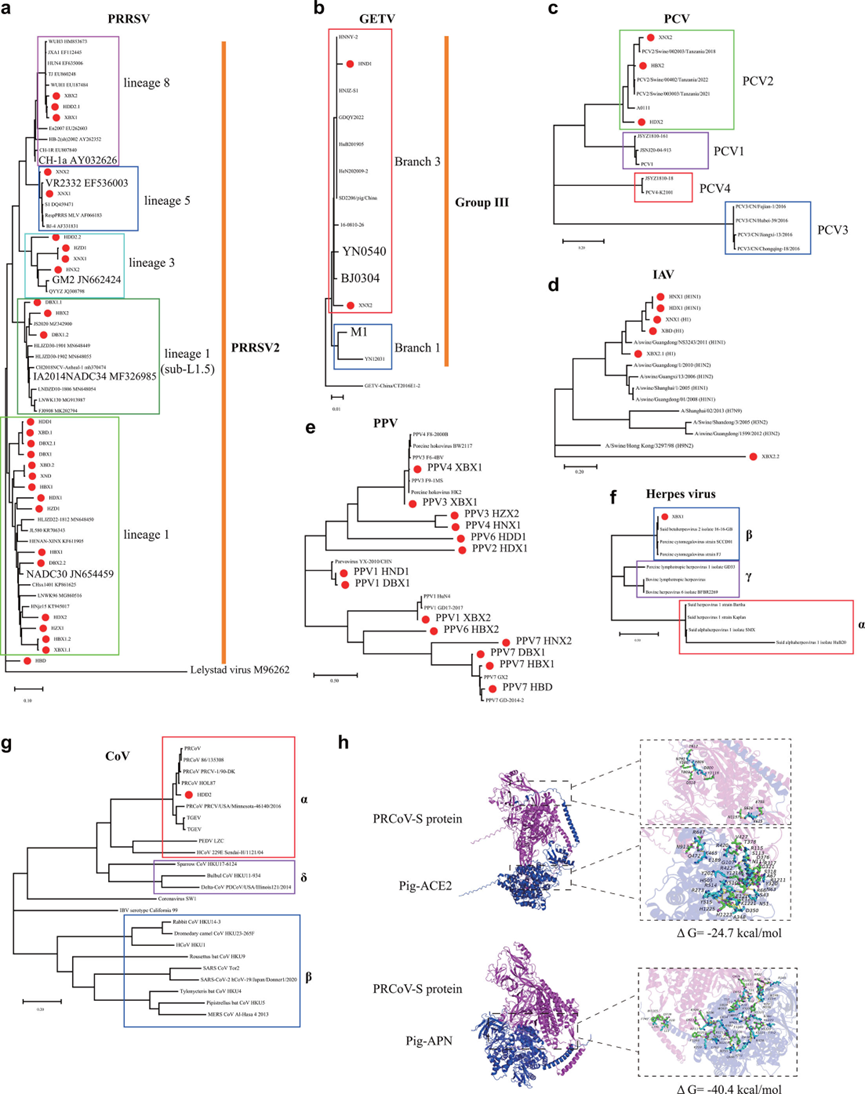
图2本研究中确定的不同病毒系统发育树
(a)猪繁殖与呼吸系统综合征(PRRSV)毒株的系统发育关系。本研究中鉴定的PRRSV菌株用小红圈表示。树是根据完整的基因组序列生成的。PRRSV1指PRRSV基因型1,而PRRSV2指PRRSV遗传型Ⅱ。
(b)Getah病毒(GETV)株系的进化关系。本研究中鉴定的GETV菌株用小红圈表示。这棵树是根据完整的基因组序列生成的。
(c)猪圆环病毒(PCV)株系的系统发育关系。本研究中鉴定的PCV株用较小的红色圆圈表示。该树是基于完整基因组序列生成的。
(d)流感A病毒(IAV)株系的系统发育关系,本研究中鉴定的IAV株用较小的红色圆圈标记。该树是基于血凝素(HA)基因的核苷酸序列生成的。
(e)猪细小病毒(PPV)株系的系统发育关系本研究中鉴定的PPV毒株用较小的红色圆圈表示。该树是基于完整基因组序列构建的。
(f)猪疱疹病毒毒株的进化关系。本研究中鉴定的疱疹病毒毒株用较小的红色圆圈表示,该树是基于完整基因组序列构建。
(g)冠状病毒(CoV)毒株的进化关系。本研究中识别的猪呼吸道冠状病毒(PRCoV)毒株用较小的红色圆圈表示。该树是基于刺突(S)基因的核苷酸序列生成的。(h)PRCoV毒株HDD2的刺突蛋白与猪ACE2蛋白和猪APN蛋白进行分子对接。
(b)Getah病毒(GETV)株系的进化关系。本研究中鉴定的GETV菌株用小红圈表示。这棵树是根据完整的基因组序列生成的。
(c)猪圆环病毒(PCV)株系的系统发育关系。本研究中鉴定的PCV株用较小的红色圆圈表示。该树是基于完整基因组序列生成的。
(d)流感A病毒(IAV)株系的系统发育关系,本研究中鉴定的IAV株用较小的红色圆圈标记。该树是基于血凝素(HA)基因的核苷酸序列生成的。
(e)猪细小病毒(PPV)株系的系统发育关系本研究中鉴定的PPV毒株用较小的红色圆圈表示。该树是基于完整基因组序列构建的。
(f)猪疱疹病毒毒株的进化关系。本研究中鉴定的疱疹病毒毒株用较小的红色圆圈表示,该树是基于完整基因组序列构建。
(g)冠状病毒(CoV)毒株的进化关系。本研究中识别的猪呼吸道冠状病毒(PRCoV)毒株用较小的红色圆圈表示。该树是基于刺突(S)基因的核苷酸序列生成的。(h)PRCoV毒株HDD2的刺突蛋白与猪ACE2蛋白和猪APN蛋白进行分子对接。
2.2、164种细菌检出,常见致病菌与“意外发现”并存
除病毒外,研究还鉴定出164种细菌,其中多种是已知的猪呼吸道疾病致病菌,同时也发现了两类此前未被重点关注的细菌:
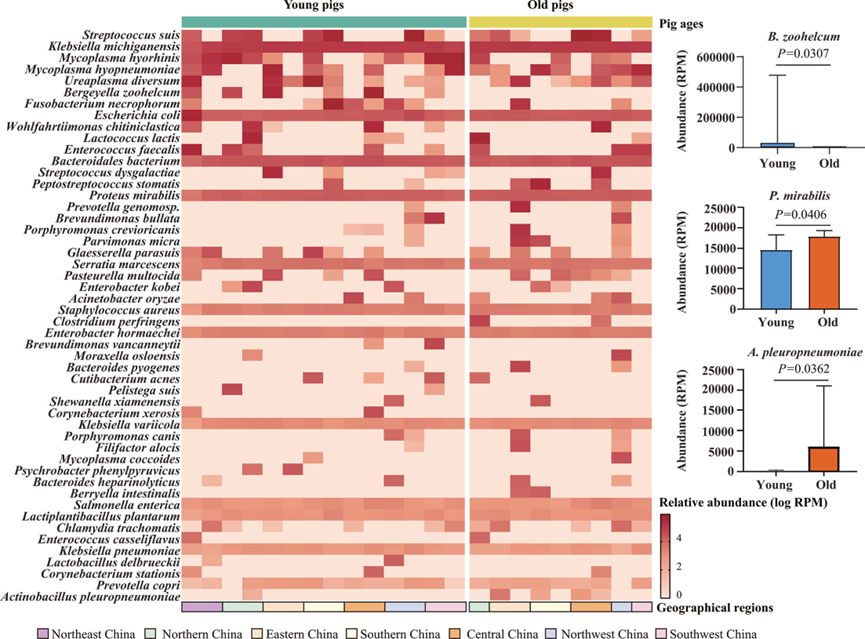
图3细菌组热图
热图显示猪肺中不同细菌种类的相对丰度,相对丰度值用对数RPM表示。用柱状图显示老龄猪和幼龄猪肺中动物性贝氏杆菌、奇异变形杆菌和胸膜肺炎放线杆菌的丰度。P值是通过曼-惠特尼U试验确定的,以进行组间多次比较。数据表示平均值土标准差。
2.2.1、主流致病菌:丰度高、分布广
猪链球菌(S.suis)是检出丰度最高的细菌,其次是猪鼻支原体(M.hyorhinis)、猪肺炎支原体(M.hyopneumoniae)、副猪嗜血杆菌(G.parasuis)和多杀巴氏杆菌(P.multocida)——这5种细菌是我国猪场每年检出率最高的呼吸道致病菌,本次研究再次确认其核心致病地位。
2.2.2、年龄差异:幼猪与成年猪的细菌谱不同
统计分析显示,幼猪肺部的伯氏杆菌(Bergeyella zoohelcum)丰度显著高于成年猪(P=0.0307),而成年猪肺部的奇异变形杆菌(Proteus mirabilis,P=0.0406)和胸膜肺炎放线杆菌(A.pleuropneumoniae,P=0.0362)丰度更高。这一差异提示,针对不同年龄段猪的呼吸道疾病,细菌防控需采取差异化策略。
2.2.3、“意外发现”:大肠杆菌与肺炎克雷伯菌的高检出
研究在病猪肺部高频检出大肠杆菌(E.coli)和肺炎克雷伯菌(K.pneumoniae)——这两种细菌此前更多被视为肠道致病菌,但本次高检出率暗示它们可能参与猪呼吸道疾病的发生(需进一步实验验证)。更值得警惕的是,二者均为人兽共患病菌,且是耐药基因的“重要载体”,其在猪呼吸道中的存在可能加剧耐药基因向人类环境扩散的风险。此外,条件性致病菌除以上两个检测出外,还有粪肠球菌(E faecalis)、金黄色葡萄球菌(S aureus),
2.3、病原体“抱团作案”:共感染是疾病加重的关键
猪呼吸道疾病并非单一病原体所致,而是“病毒×病毒”、“病毒×细菌”、“细菌×细菌”共感染的结果。研究通过Spearman相关性分析,绘制出病原体相互作用网络,揭示了三大核心关联:
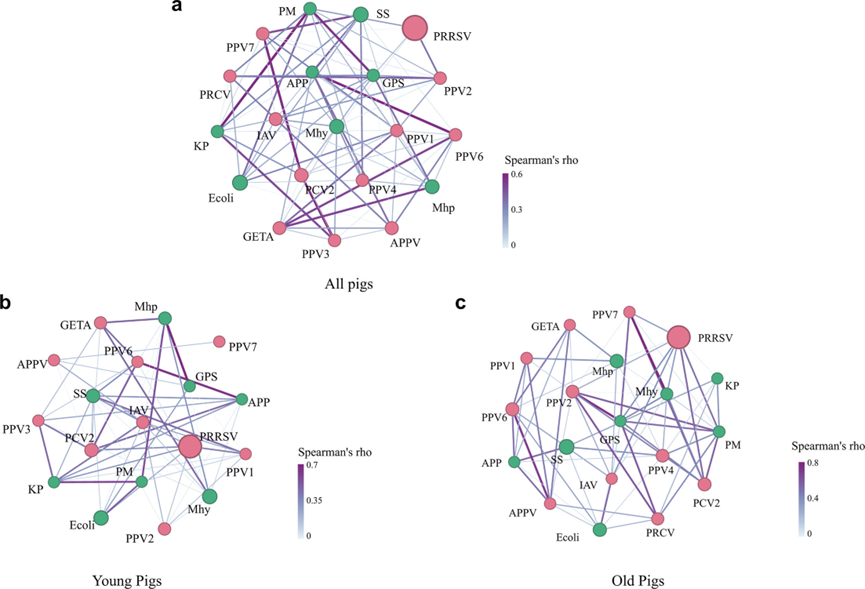
图4不同病原微生物的相关性。
(a)患有呼吸系统疾病的所有猪的肺内所鉴定的不同病原微生物之间的相互关系。
(b)患有呼吸系统疾病的幼猪肺内所鉴定的不同病原微生物之间的相互关系。
(c)在患有呼吸道疾病的成年猪肺部发现的不同致病微生物之间的相关性。
相关网生成的Spearman的相关分析的基础上。病毒被描绘成粉红色的节点,而细菌被描绘为绿色的节点。每个结点的大小对应于各自病原体的相对丰度,每个边缘的厚度和颜色强度与Spearman的rho值成正比。APP,胸膜肺炎放线杆菌;APPV,非典型猪瘟病毒;Ecoli,大肠杆菌;GETA,Getah病毒;GPS,副猪嗜血杆菌;IAV,甲型流感病毒;KP肺炎克雷伯菌;Mhp,猪肺炎支原体;Mhy,猪鼻支原体;PCV2、猪圆环病毒2;PM,多杀性巴氏杆菌;PPV1、猪细小病毒1;PPV2、猪细小病毒2;PPV3、猪细小病毒2:PPV4、猪细小病毒4;PPV6、猪细小病毒6:PPV7、猪细小病毒7:PRCV,猪呼吸道冠状病毒:PRRSV、猪生殖和呼吸综合征病毒:SS,猪链球菌。
(b)患有呼吸系统疾病的幼猪肺内所鉴定的不同病原微生物之间的相互关系。
(c)在患有呼吸道疾病的成年猪肺部发现的不同致病微生物之间的相关性。
相关网生成的Spearman的相关分析的基础上。病毒被描绘成粉红色的节点,而细菌被描绘为绿色的节点。每个结点的大小对应于各自病原体的相对丰度,每个边缘的厚度和颜色强度与Spearman的rho值成正比。APP,胸膜肺炎放线杆菌;APPV,非典型猪瘟病毒;Ecoli,大肠杆菌;GETA,Getah病毒;GPS,副猪嗜血杆菌;IAV,甲型流感病毒;KP肺炎克雷伯菌;Mhp,猪肺炎支原体;Mhy,猪鼻支原体;PCV2、猪圆环病毒2;PM,多杀性巴氏杆菌;PPV1、猪细小病毒1;PPV2、猪细小病毒2;PPV3、猪细小病毒2:PPV4、猪细小病毒4;PPV6、猪细小病毒6:PPV7、猪细小病毒7:PRCV,猪呼吸道冠状病毒:PRRSV、猪生殖和呼吸综合征病毒:SS,猪链球菌。
2.3.1、PRRSV是“共感染核心”
PRRSV与多种病毒(如GETV、PPV2)和细菌(如猪链球菌、多杀巴氏杆菌、肺炎克雷伯菌、猪鼻支原体、猪肺炎支原体、副猪嗜血杆菌)均存在显著正相关——这意味着感染PRRSV的猪,更易同时感染其他病原体,从而加重病情。
2.3.2、新兴病毒与细菌的关联
GETV常与猪肺炎支原体、副猪嗜血杆菌、胸膜肺炎放线杆菌及PPV1/PPV6共感染;流感病毒(IAV)则与胸膜肺炎放线杆菌、副猪嗜血杆菌存在关联,这些共感染组合可能是特定地区猪呼吸道疾病爆发的“诱因”。
2.3.3、大肠杆菌与肺炎克雷伯菌的协同作用
尽管二者在猪呼吸道疾病中的作用尚不明确,但研究发现它们与多杀巴氏杆菌存在强相关性——这提示三者可能通过协同作用加剧肺部损伤。
2.4、五年暗战:病原的“进化诡计”
研究人员还对NCBI中2017-2023年间的主要病原基因组序列进行了回顾性分析,以了解其流行趋势。
2.4.1、病毒变形记
对PRRSV的分析显示,PRRSV2型仍然是绝对优势型别(97.06%),只有少量PRRSV1型(2.94%)。PRRSV2中毒株包括L1、L3、L5和L8谱系,2021年L8分支占58%,但2023年L1分支占62%,也是最近疫苗失效的主要原因。
对IAV的分析表明,H1N1是2009年后的主要血清型,但其他血清型:如H3N8、H10N5和近期的H7N9也有检测,其中H7N9禽源毒株现身华中地区猪场。
PCV回顾分析显示,虽然PCV2仍是主要类型,但PCV3的流行率在2022年后有所增加,PCV4也在中国猪场中存在,三年的检出率飙升了17倍。
PPV的流行情况复杂,PPV1在近年来占主导地位。所有来自中国猪的GETV毒株均属于GIII型。
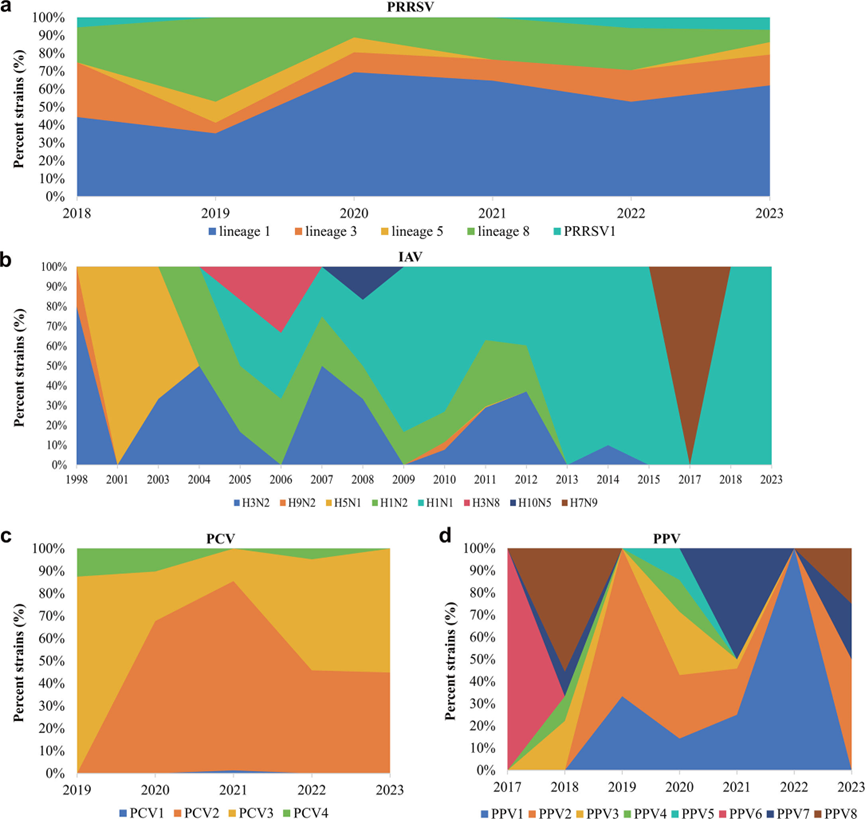
图5主要病毒种类的变化情况
(a)根据NCBI公布的基因组序列,回顾性地确定了2018年至2023年中国猪场猪繁殖与呼吸系统综合征(PRRS)的动态变化。
(b)根据NCBI公布的基因组序列,回顾性确定1998年至2023年中国养猪场甲型流感病毒(IAV)的变化动态。
(c)根据NCBI中已发表的基因组序列,回顾性地确定2019年至2023年期间中国猪场猪圆环病毒(PCV)的动态变化。
(d)根据NCBI中已发表的基因序列,回顾性地确定2017年至2023年期间中国养猪场猪细小病毒(PPV)的动态变化。
(b)根据NCBI公布的基因组序列,回顾性确定1998年至2023年中国养猪场甲型流感病毒(IAV)的变化动态。
(c)根据NCBI中已发表的基因组序列,回顾性地确定2019年至2023年期间中国猪场猪圆环病毒(PCV)的动态变化。
(d)根据NCBI中已发表的基因序列,回顾性地确定2017年至2023年期间中国养猪场猪细小病毒(PPV)的动态变化。
2.4.2、细菌血清型暗战
猪链球菌:2型是全国主流血清型,但东北地区的9型检出率明显高于其他地区——这意味着东北地区猪场需重点关注9型疫苗的研发或应用。
副猪嗜血杆菌:4型和5型是全国主导血清型,但2023年除东北地区外,14型在其他地区新兴流行——提示需更新疫苗组分以覆盖14型。
多杀巴氏杆菌:A群和D群是主流,且A群丰度高于D群;此外,华东和华中地区检出F群——F群巴氏杆菌此前罕见于猪场,但具有高致病性,需针对性防控。
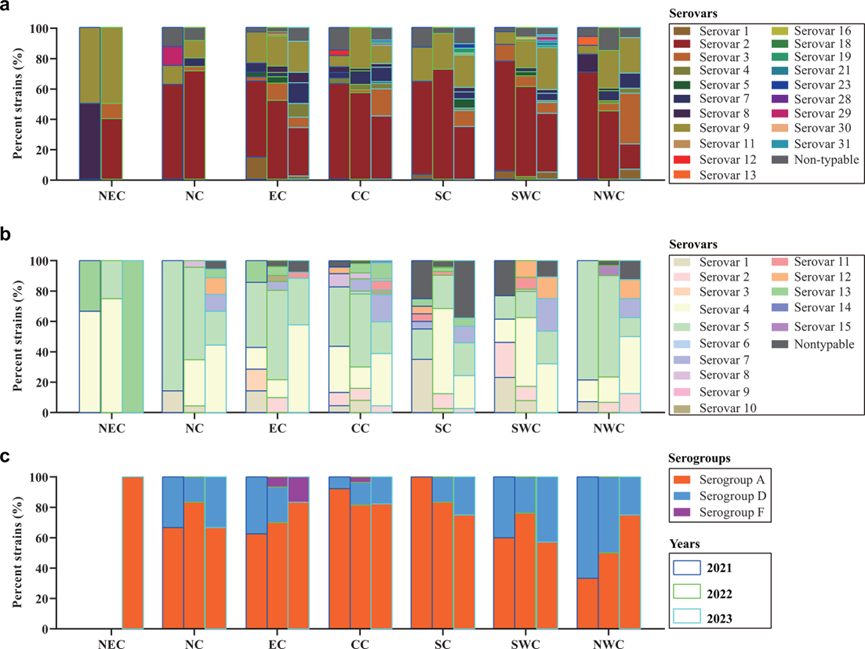 图6主要细菌血清型分布情况。
图6主要细菌血清型分布情况。 (a)2021年至2023年与猪呼吸系统疾病相关的不同血清型猪链球菌在中国不同地区的分布。
(b)2021-2023年中国不同地区与猪呼吸系统疾病相关的副猪链球菌不同血清型的分布情况。
(c)2021-2023年中国不同地区与猪呼吸系统疾病相关的多杀性巴氏杆菌不同血清型的分布情况。
NEC,中国东北部;EC,中国东部;NC,中国北部;CC,中国中部;SC,中国南部;SWC,中国西南部;NWC,中国西北部。
(b)2021-2023年中国不同地区与猪呼吸系统疾病相关的副猪链球菌不同血清型的分布情况。
(c)2021-2023年中国不同地区与猪呼吸系统疾病相关的多杀性巴氏杆菌不同血清型的分布情况。
NEC,中国东北部;EC,中国东部;NC,中国北部;CC,中国中部;SC,中国南部;SWC,中国西南部;NWC,中国西北部。
2.5、48种耐药基因检出,移动元件加剧扩散风险
抗生素滥用导致的细菌耐药性,是全球养猪业面临的另一挑战。研究在病猪肺部鉴定出48种耐药基因(ARGs),可抵抗13类抗生素,且存在三大风险特征:
耐药谱集中:四环素类耐药基因丰度最高,51%菌株携带tet耐药基因,其次是大环内酯-林可酰胺-链阳菌素B(MLS)类和氨基糖苷类——这三类抗生素是猪场治疗呼吸道疾病的常用药,高耐药基因丰度反映了临床滥用问题。其次还检测到负责介导对消毒剂耐药的基因(如leuO)
多耐药基因存在:检出efrA、efrB、evgS等基因,可同时抵抗氟喹诺酮类、大环内酯类等多种抗生素,其中efrA基因可同时抵抗3类抗生素(氟喹诺酮+大环内酯+利福平),这类基因的存在会导致“无药可用”的困境。
与移动遗传元件(MGEs)关联紧密:多数耐药基因与转座子(如tnpAIS1、ISCau1)和质粒(如Col(BS512)、Col(YC))显著相关,这些移动元件可在不同细菌间“跳跃”,导致耐药基因快速扩散,增加了这些耐药基因传播的风险,甚至传播给人源细菌。
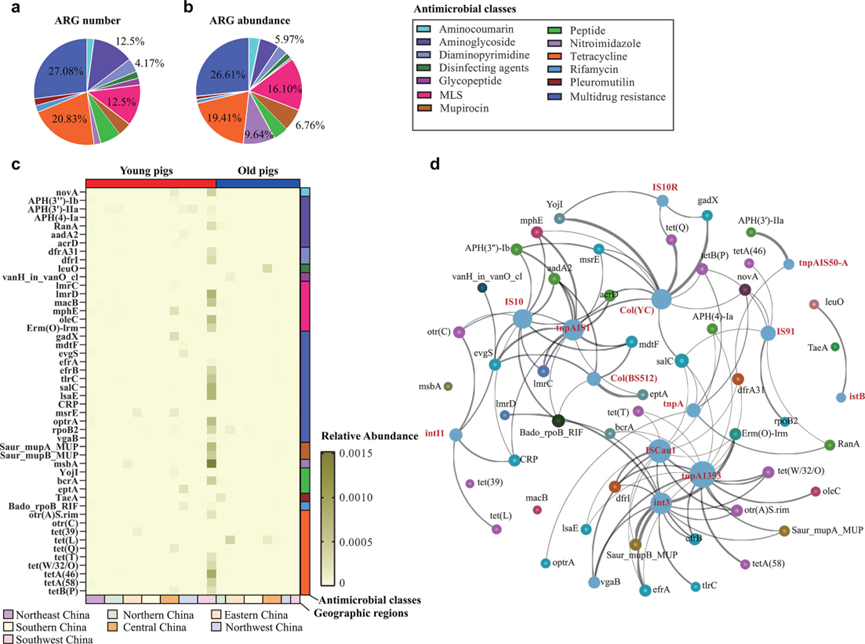
图7抗菌素耐药基因(ARG)及其与可移动遗传元件(MGES)的关系
(a)一个扇形图,显示对不同抗菌素类产生耐药性的ARG分布情况。
(b)一个扇形图,显示了赋予对不同抗菌素类耐药性的ARG的丰度。
(c)显示中国不同地区患有呼吸系统疾病的猪肺中不同ARG丰度的热图。
(d)中国呼吸系统疾病猪肺中ARGS和MGEs的相关性网络图。网络图的生成基于皮尔逊相关分析。每个节点的大小与连接的数量成正比,每个边缘的厚度与皮尔逊的rho值成正比。ARG的标签是黑色的,而MGE的标签是红色的。
3、讨论
本次全国性研究不仅揭示了猪呼吸道疾病的病原体“全景”,更从流行病学、疾病防控和公共卫生角度,为行业提供了关键参考。
3.1、核心病原体的防控需“动态调整”
3.1.1、PRRSV:长期主导,谱系需重点监测
PRRSV自1996年在我国首次报道以来,始终是养猪业的“头号敌人”。本次研究确认PRRSV2型L1谱系(NADC-30/34株)为当前主流,提示疫苗研发需以L1谱系为核心,同时警惕其他谱系(如L3、L5)的流行。此外,PRRSV与其他病原体的共感染特性,意味着防控不能仅针对PRRSV,还需结合其他病原体的监测。
3.1.2、PCV3/PCV4:新兴威胁,需持续追踪
尽管本次研究仅在肺组织中检出PCV2,但回顾性分析显示PCV3在2022年后流行率上升,PCV4也已出现。已知PCV3/4可导致猪呼吸道损伤外的其他症状(如繁殖障碍),其潜在危害可能被低估,未来需建立专项监测体系。
3.1.3、GETV与冠状病毒:人兽共患病风险需警惕
GETV作为人兽共患病病毒,其在猪呼吸道中的发现,提示需警惕“蚊-猪-人”的传播链;而新型冠状病毒HDD2的“双靶点”结合能力,意味着它可能具有更强的致病性和传播性,需进一步研究其对猪和人类的威胁。
3.2、细菌防控的“难点”:血清型复杂与耐药风险
3.2.1、血清型地区分化:疫苗需“定制化”
猪链球菌、副猪嗜血杆菌等细菌的血清型存在显著地区差异(如东北猪链球菌9型、华东多杀巴氏杆菌F群),传统“一刀切”的疫苗可能无法覆盖所有流行血清型。未来需推动“区域性疫苗”研发,或开发多血清型联合疫苗。
3.2.2、大肠杆菌与肺炎克雷伯菌:不可忽视的“隐形风险”
二者在病猪肺部的高检出,不仅可能参与呼吸道疾病,更作为耐药基因载体,加剧人兽共患病耐药风险。这提示猪场需减少不必要的抗生素使用,同时加强对这两种细菌的监测,避免其在猪群中定植扩散。
3.3、共感染机制:未来研究的“核心方向”
本次研究证实共感染是猪呼吸道疾病加重的关键,但多数病原体间的相互作用机制仍不明确(如PRRSV如何促进猪链球菌感染)。未来需深入研究共感染的分子机制,例如:病毒是否通过破坏呼吸道黏膜屏障,为细菌入侵创造条件?细菌是否通过分泌毒素,增强病毒的复制能力?这些研究将为开发“针对共感染的防控策略”提供依据。
3.4、耐药性防控:需从“源头减量”到“监测阻断”
减少抗生素滥用:四环素类、MLS类抗生素的高耐药基因丰度,反映了临床滥用问题。猪场需优化用药方案,例如基于药敏试验精准用药,或采用益生菌、噬菌体等非抗生素替代方案。
阻断耐药基因扩散:耐药基因与移动元件的关联,意味着需加强对猪场环境(如粪便、污水)的处理,防止耐药基因通过环境传播给人类细菌。同时,需建立全国性的猪源细菌耐药监测网络,实时掌握耐药谱变化。
3.5、研究局限性与未来方向
本次研究虽覆盖全国25个省份,但仍存在不足:受非洲猪瘟生物安全限制和新冠疫情影响,样本量未能进一步扩大;采用“样本pooling测序”可能影响病原体定量的准确性。未来研究需:1)扩大样本量,覆盖更多地区和猪场类型;2)采用单样本测序,提高定量准确性;3)结合动物实验,验证新兴病原体(如GETV、HDD2)的致病性;4)深入研究共感染机制和耐药基因传播路径。
4、总结
为猪呼吸道疾病防控“点亮灯塔”
这项全国性研究首次通过宏转录组与宏基因组联合技术,绘制了我国猪呼吸道疾病的“病原体-耐药基因”全景图,其核心价值在于:
填补认知空白:明确了21种病毒、164种细菌的流行特征,发现GETV、新型冠状病毒等新兴病原体,为病因诊断提供依据;
指导疫苗研发:揭示主要病原体的谱系/血清型分布(如PRRSV2型L1谱系、猪链球菌2型),为“定制化疫苗”提供数据;
预警公共卫生风险:指出GETV的人兽共患病可能、大肠杆菌/肺炎克雷伯菌的耐药基因载体作用,为跨物种疾病防控提供参考;
优化防控策略:强调共感染的重要性和耐药性的危害,推动从“单一病原体防控”向“综合管理”转变。
对于养猪业而言,这项研究不仅是一次“病因普查”,更是未来防控工作的“灯塔”,只有基于精准的病原体监测、动态的谱系/血清型追踪、科学的抗生素使用,才能真正攻克猪呼吸道疾病这一“顽疾”,保障产业健康发展与食品安全。
注:以上数据仅供参考,不作任何投资建议
















